“Nuestro método para predecir estructuras de complejos moleculares entre proteínas y fármacos (2Vinardo) resultó el mejor en su categoría entre los 34 grupos de todo el mundo que participaron de la competencia internacional CASP16”, comentó en su cuenta de la red social X Rodrigo Quiroga, quien junto a Marcos Villarreal desarrolló esta novedad desde el Departamento de Química Teórica y Computacional de la Facultad de Ciencias Químicas, en la Universidad Nacional de Córdoba (UNC).
Desde 1994, cada 2 años, el certamen Critical Assessment of Structure Prediction (CASP) –Evaluación Crítica de la Predicción de Estructuras- reúne a grupos de todo el mundo que se dedican a competir en distintos ejercicios de predicción de estructuras de proteínas, utilizando diversos métodos computacionales.
El evento es uno de los más convocantes de la especialidad y en ediciones anteriores contó, por ejemplo, con la participación de los actuales Premio Nobel de Química, cuyos programas también formaron parte de la última competencia.
Los métodos que emplean Inteligencia Artificial (IA) vienen cobrando protagonismo en este campo. Las predicciones de Alphafold -el método computacional de aprendizaje profundo basado en IA, cuyos desarrolladores ganaron el Premio Nobel de Química 2024- tuvieron gran éxito durante las ediciones 2020 y 2022 de CASP.
Sin embargo, una de las particularidades del desarrollo de Quiroga y Villarreal es la combinación de métodos de entrenamiento utilizados en aprendizaje profundo (IA) con técnicas tradicionales, buscando potenciar las ventajas de ambos para encontrar nuevas alternativas destinadas al diseño de fármacos.
“Con nuestro algoritmo más simple y en términos computacionales más económico, pudimos ganarles a métodos que usan Alphafold 2 o Alphafold 3. No quiere decir que siempre sea así, pero significa que los métodos más tradicionales aún pueden competir y tienen múltiples ventajas por sobre los basados en IA”, señaló Quiroga.
En la sección L1000 de CASP, la metodología desarrollada en la UNC se impuso a equipos de prestigiosas universidades estadounidenses como las de Boston, Missouri, Washington y Pittsburgh; la Universidad Nacional de Seúl (Corea del Sur) y las de Huazhong y Jiangsu (China), cuyos científicos cuentan con presupuestos anuales para investigación que pueden llegar a ser hasta mil veces superiores a los de Argentina.

2Vinardo, “hecho en la UNC”
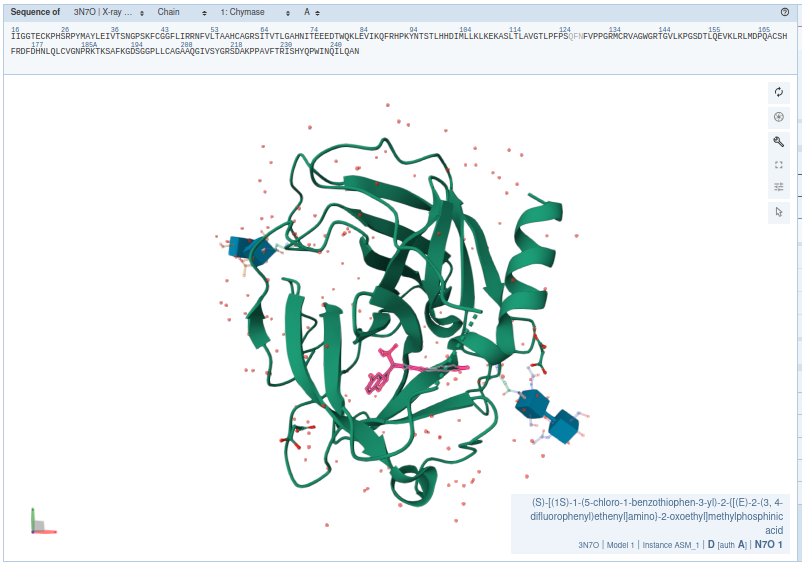
En el certamen internacional, 2Vinardo resultó el método que predijo estructuras más similares a las experimentales. “Nuestro método se llama 2Vinardo porque es el heredero de Vinardo, que a su vez está basado en Autodock Vina, el programa de acoplamiento molecular más popular y usado del mundo”, explicó Quiroga.
Se trata de una función de puntuación empírica o tradicional, en la que cada simulación de interacción entre un fármaco y una proteína resulta extremadamente veloz (menos de 1 segundo en promedio en una computadora común o de escritorio). A diferencia de esto, los métodos basados en IA pueden demorar desde 30 segundos hasta 3 minutos por cada par proteína-ligando (molécula orgánica pequeña).
La velocidad no es un aspecto menor en el campo de las simulaciones. En el caso de 2Vinardo resulta fundamental al llevar adelante el proceso conocido como cribado virtual o virtual screening, a través del cual se ejecutan millones de simulaciones para encontrar compuestos capaces de inhibir una enzima de interés médico.
Por otro lado, las predicciones de los métodos tradicionales se destacan porque resultan más fáciles de interpretar, mientras que los resultados alcanzados a través de IA siguen generando incertidumbre en la comunidad científica. “Son métodos de caja negra, no podemos saber por qué predicen lo que predicen. Los métodos empíricos están basados en la físico-química y nos permiten analizar por qué obtenemos ciertos resultados y por qué el método a veces se equivoca logrando intuir, de ese modo, cómo mejorarlo”, sintetizó Quiroga.
Debido a esto, el método desarrollado en los laboratorios de la UNC combina las ventajas de las funciones de puntuación tradicionales con los de optimización de parámetros empleados en el aprendizaje profundo mediante IA.

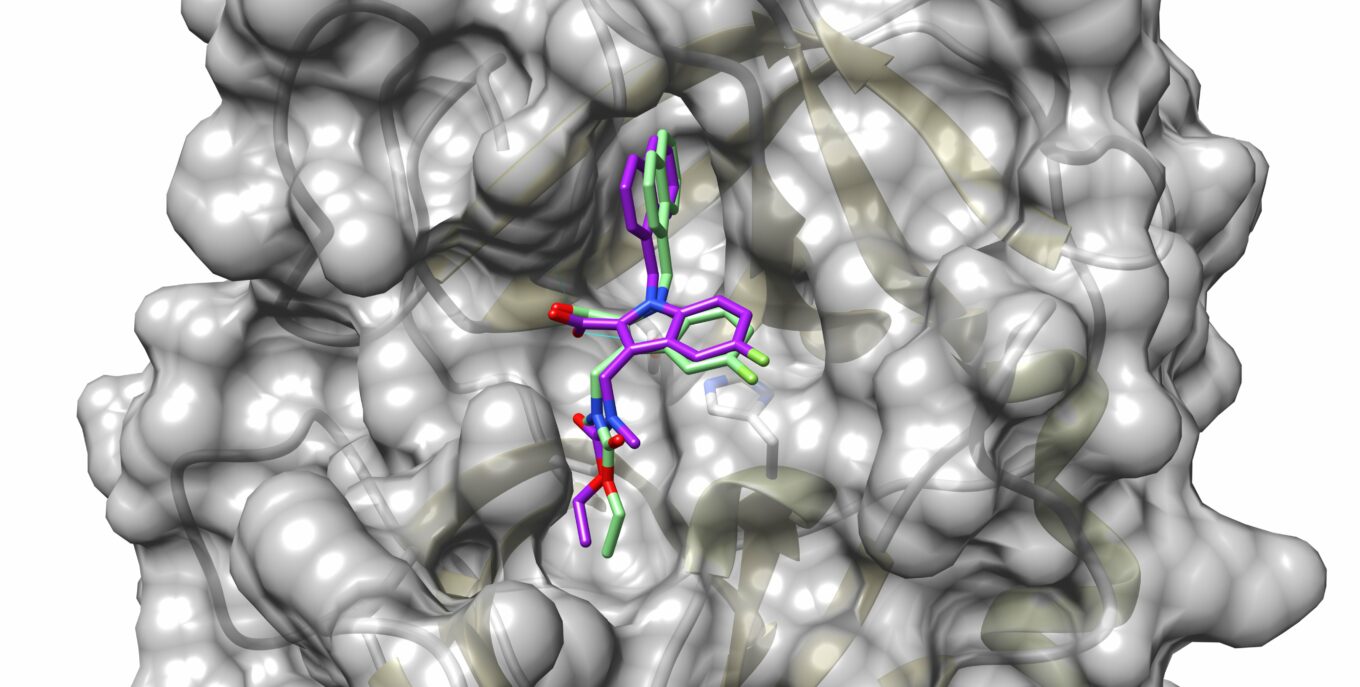
Según el docente, “esto va a contramano de las tendencias, pero las funciones de puntuación empíricas o tradicionales en algunos casos demuestran tener similares o mejores resultados que los métodos basados en IA más novedosos”.
Desde 2015, el trabajo de estos científicos cordobeses tiene como objetivo desarrollar simulaciones computacionales que permitan predecir dónde, cómo y con qué energía interaccionan los fármacos y las proteínas. Posteriormente, estos métodos pueden utilizarse para diseñar o descubrir nuevos fármacos destinados, por ejemplo, a enfermedades que aún no tienen un tratamiento efectivo. Luego, estas predicciones computacionales necesitan ser confirmadas de manera experimental. En general, permiten reducir el tiempo y el dinero invertido hasta en un 99%.
En el caso de Quiroga y Villarreal, esos desarrollos son llevados a cabo con equipos de la FCQ, del Instituto de Investigaciones en Físico-Química de Córdoba (INFIQC-CONICET) y las supercomputadoras de la UNC, como Serafín, que se encuentran en el Centro de Computación de Alto Desempeño (CCAD), del cual la Facultad forma parte.
“Es un orgullo lograr estos resultados en un contexto de desfinanciamiento de las universidades públicas nacionales y CONICET. Aun con un presupuesto ínfimo, resulta increíble que la ciencia argentina sea competitiva a nivel mundial. Ojalá podamos lograr detener la destrucción del todavía excelente sistema científico nacional”, sostuvo Quiroga.



